Leggendo la notizia pubblicata su vari quotidiani on line il primo aprile ho pensato al famoso “pesce”… come si può esultare dicendo che abbiamo la mappa del genoma umano? Facendo un rapido calcolo erano trascorsi almeno ventina di anni (per la precisione era l’inizio del 2001) da quando era stato annunciato al mondo, in contemporanea su Nature1 e su Science2, che avevamo sequenziato il genoma umano!
Dopo una prima lettura ai titoli ho pensato quindi che il punto fosse che, finalmente, fossimo riusciti a capire a cosa serve tutta quella parte del genoma, quantitativamente non indifferente, inizialmente definita “spazzatura” e che invece col tempo stiamo scoprendo che serve a regolatore tutta una serie di meccanismi biologici… e quindi ho cercato spasmodicamente, dal telefonino, l’articolo di riferimento su Science, dal titolo tanto asciutto quanto eloquente:
“The complete sequence of the human genome3“, tradotto letteralmente “la sequenza (completa) del genoma umano”.

E invece no, per capire il significato di quel che abbiamo mappato ci vorrà ancora tempo, mentre invece è stata pubblicata una mappa aggiornata: scopriamo (anzi ci ricordano) che ne mancava circa un 8% di “materiale” da sequenziare, che non è poco.
A leggere meglio qualche dettaglio ci manca ancora, in particolare sul cromosoma Y (per chi non può attendere un commento su questo suggerisco il “post scriptum” alla fine dell’articolo).
La fine dell’inizio
Ma eravamo già consapevoli di questa mancanza? In realtà si (e qui faccio un mea culpa), e in effetti si sono susseguite diverse “versioni” del genoma umano negli ultimi anni. Ad esempio già nel 2004 erano stati pubblicati diversi articoli, alcuni anche di facile lettura, tra cui uno eloquente: “End of the beginning4“, la fine dell’inizio, in cui si asseriva che, rispetto alle “bozze” pubblicate con enorme enfasi ad inizio 2001, mancava ancora circa un 10% di eucromatina e 30% DNA totale (comprensivo anche della cosiddetta “eterocromatina“).
Per non addentrarci troppo nella questione possiamo dire che la eucromatina è la porzione del genoma (diviso nei vari cromosomi) che è attiva per la trascrizione, l’eterocromatina è la parte non attiva5.

Ma perché ci sono parti attive e parti non attive? Basterà pensare che il nostro DNA (diviso in 2 coppie di 23 cromosomi) è lungo circa 2 metri e va impacchettato, per ogni cellula, in ogni spazio minuscolo, qualcosa come 6 millesimi di millimetro (le dimensioni di una cellula umana media), e a voler essere precisi in una zona ben circoscritta chiamata nucleo6. Inoltre, non deve stare li “fermo” ma deve poter essere utilizzato per trascrivere i geni di interesse e, all’occorrenza e in fasi specifiche, essere copiato e duplicato completamente.
Quindi il DNA, all’interno del nucleo, è in buona parte in una conformazione estremamente compatta e non può essere trascritto (eterocromatina) e ci sono solo alcune porzioni in cui è più “rilassato” e pronto per essere trascritto (eucromatina). Il tutto, come dicevamo, in relazione allo stato vitale della cellula e alla sua tipologia.
Per capirci meglio, so che può sembrare banale per molti di voi, una cellula cardiaca e una cellula delle isole di Langerhans (pancreas) hanno lo stesso identico DNA, ma zone attive (e geni espressi) completamente diverse!
Ma torniamo al “buco”
Ma cosa c’entra tutto questo con i buchi che avevamo su queste zone?
Vent’anni fa non avevamo la tecnologia per sequenziare lunghi frammenti di DNA (ed eravamo anche molto più indietro con la bioinformatica), pertanto – tra le varie strategie – ed in una competizione “sana” tra pubblico e privato si era sviluppata ed utilizzata la tecnologia “BAC”, acronimo di Bacterial Artificial Chromosome.
Per sequenziare il DNA avevamo bisogno di molte copie degli stessi frammenti relativamente corti e non si potevano ottenere facilmente con una PCR, pertanto sono stati creati dei cromosomi artificiali che poi venivano inseriti all’interno di un batterio “ingannandolo” per fargli ritenere che fosse roba sua. Così i famosi Escherichia coli facevano il lavoro sporco per noi.

Ma, come tutte le cose “facili”, c’erano degli inghippi:
- Il genoma sequenziato non rappresentava un individuo specifico (sia per aspetti tecnici che per aspetti più “sociali”), era un mosaico di diversi individui;
- Alcune parti del genoma sono più nascoste di altre (i diversi aspetti della cromatina) e i telomeri sono in buona parte in “formazione” di eterocromatina;
- Mettere insieme i pezzi di un puzzle fatto di decine di migliaia di pezzi è decisamente più complesso e a rischio di errore rispetto allo stessa immagine composta da pezzi più grandi.
Quando le diverse parti sono state assemblate non tutti i pezzi combaciavano e si è dovuto lasciare qualche spazio bianco o forzare un pò l’accoppiamento.
I telomeri
Quel che abbiamo oggi è il risultato del consorzio T2T (da un telomero all’altro), quindi la sequenza dei cromosomi completi7.
In realtà, semplicemente andando a “guardare le figure” ci sono centinaia di spunti di riflessione. Abbiamo informazioni appunto sulle zone “nascoste” del DNA, sulla metilazione (che ha proprio effetti regolatori sulla trascrizione del DNA e che dipende anche dall’ambiente che ci circonda), su una serie di aspetti evolutivi…
Nei prossimi giorni leggerò meglio, qui però mi concentro solo sul titolo e cioè sui telomeri.

I telomeri, detta facile, sono la parte finale dei cromosomi, che non fanno cose particolarmente importanti… tant’è che vengono “consumati”, un pezzetto alla volta, ogni volta che avviene una replicazione della cellula. O meglio, questa cosa avverrebbe se non ci fosse un enzima chiamato telomerasi, che ha il compito, per così dire, di riattaccare dei pezzettini ogni volta.
Quando le telomerasi non funzionano bene (per motivi indotti dall’ambiente o genetici) si possono innescare tutta una serie di malattie, a partire da aumentato rischio di sviluppare cancro, fino ad arrivare all’invecchiamento precoce.

Ora, evidentemente, una trattazione completa sui telomeri esula non solo dallo spazio su questo blog, dalla vostra pazienza e soprattutto dalle mie competenze…
Accontentiamoci di ricordare che nel 2009 è stato assegnato il Nobel per la medicina a 3 scienziati (Greider, Blackbum e Szotak) proprio per i loro studi sui telomeri e le telomerasi.
Per qualche dettaglio in Italiano vi rimando ad un articolo divulgativo su “Le Scienze9” scritto nel 2009, mentre per maggiori informazioni e delle belle immagini conviene dare uno sguardo a quanto scritto sul sito web del premio Nobel.
Per chi non volesse cliccare sugli articoli indicati e/o uscire da questo blog aggiungo solo che ci sono decine e decine di malattie correlate ai “telomeri corti” e questo dovrebbe convincerci sul fatto che siano utili e che includerli nella mappa del genoma sia non solo opportuno ma necessario.
L’immagine che segue proviene da una interessantissima review del 2021 su Nature10
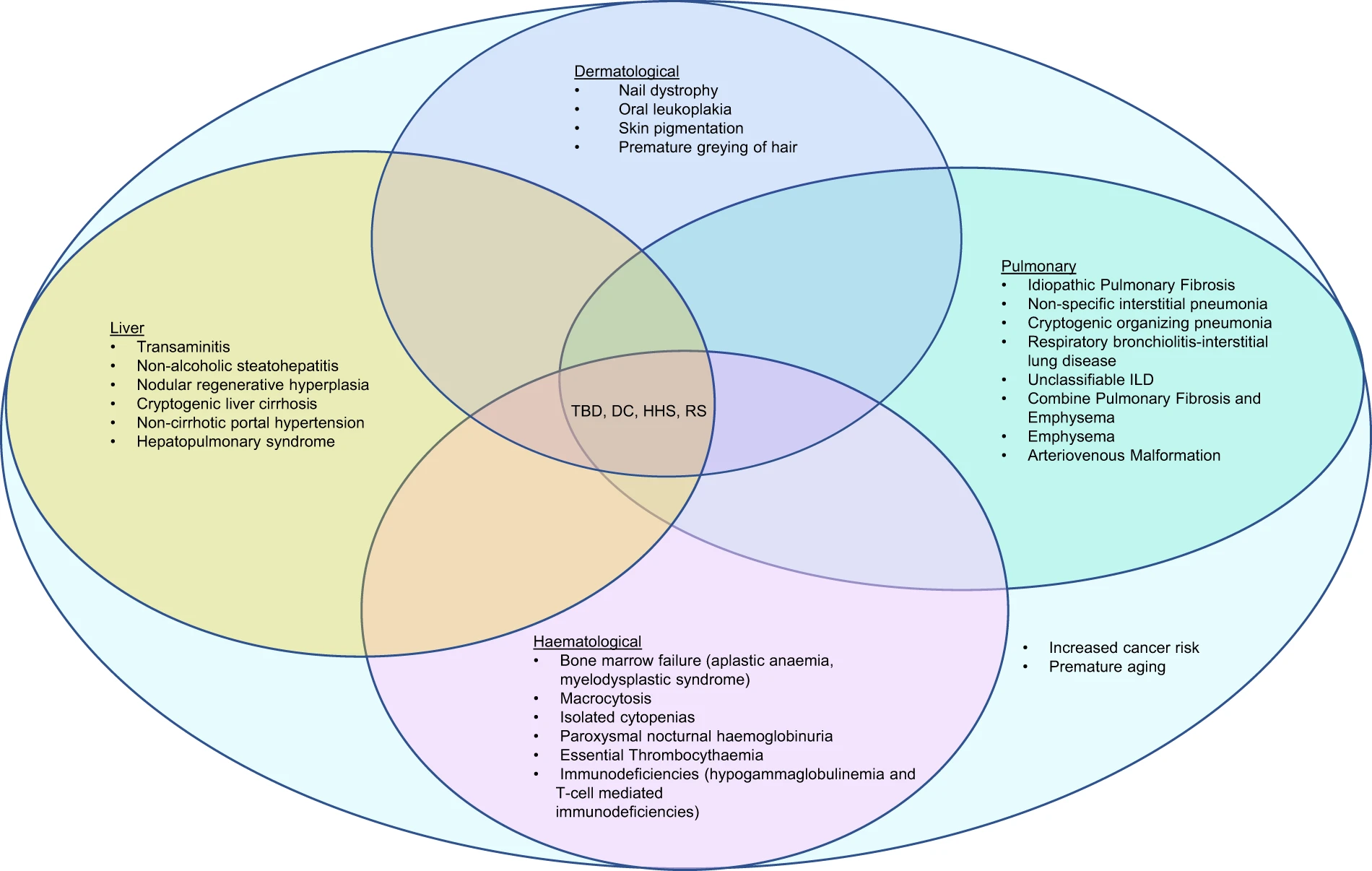
La tecnologia
Un cenno alla tecnologia. Negli ultimi anni c’è stata una enorme evoluzione dei sistemi di sequenziamento, sia in termini di qualità, semplicità d’uso, velocità. Al tempo stesso vi è stata una caduta del costo del sequenziamento. I sequenziatori di nuova generazione (Thermofisher ed Illumina) sequenziano, con alte velocità e precisione, frammenti relativamente corti (poche centinaia di basi), il che va benissimo se so cosa andare a cercare. Cosa diversa invece avviene per il sequenziamento de novo dell’intero genoma.
Qualche anno fa Oxford Nanopore e PacBio hanno dimostrato che era possibile sequenziare frammenti molto più lunghi, ma per alcuni anni il tasso di errore è rimasto abbastanza alto (superiore al 5%). Negli ultimi anni però queste tecnologie sono state enormemente affinate e siamo arrivati con PacBio a sequenziare frammenti molto più lunghi (anche fino a 20.000 paia di basi) con error rate inferiore allo 0,1%.

Conclusioni
Insomma, quello che è stato pubblicato il 31/3/2022 e comunicato il 1/4/2022 non è un pesce d’aprile, né la scoperta dell’acqua calda. Ma una mappa completa e ad altissima risoluzione.
Ricordiamoci che buona parte dei nuovi farmaci sono collegati a dei target molecolari studiati a partire dal genoma, per non parlare di tutte le attività di ingegnerizzazione del genoma (abbiamo già parlato di CRISPR) e di terapia genica.
Non parliamo pertanto di un “vezzo da addetti ai lavori”, ma uno strumento necessario agli addetti ai lavori per comprendere meglio come siamo fatti e come agire per migliorare la salute pubblica.
PS come accennavo all’inizio, in questa mappa aggiornata manca ancora qualche pezzo del cromosoma Y (e poi si dice che le donne siano poco rappresenta nel mondo scientifico)… di questo cromosoma in generale si parla con disinvoltura usando termini poco gentili, ho letto che è un “deserto evoluzionistico11” ricco di materiale genetico spazzatura e povero di geni, tra i quali comunque troviamo alcuni che sono correlati alla locomozione, alta pressione e risposta allo stress12. Ma anche di questo, forse, parleremo un’altra volta.
PPS Last buy not least: una speranza di… PACE. Leggendo le afferenze degli oltre 80 autori scopriamo che in buona parte sono americani… e russi.
Riferimenti bibliografici:
- International Human Genome Sequencing Consortium. Initial sequencing and analysis of the human genome. Nature 409, 860–921 (2001). https://doi.org/10.1038/35057062
- The Sequence of the Human Genome. Science 1304-1351 (2001). doi:10.1126/science.1058040
- The complete sequence of a human genome Science, 376 (6588), doi:10.1126/science.abj6987
- Stein, L. End of the beginning. Nature 431, 915–916 (2004). https://doi.org/10.1038/431915a
- Croken MM, Nardelli SC, Kim K. Chromatin modifications, epigenetics, and how protozoan parasites regulate their lives. Trends Parasitol. 2012 May;28(5):202-13. doi: 10.1016/j.pt.2012.02.009. Epub 2012 Apr 3. PMID: 22480826; PMCID: PMC3340475.
- Piovesan A, Pelleri MC, Antonaros F, Strippoli P, Caracausi M, Vitale L. On the length, weight and GC content of the human genome. BMC Res Notes. 2019 Feb 27;12(1):106. doi: 10.1186/s13104-019-4137-z. PMID: 30813969; PMCID: PMC6391780.
- S. J. Hoytet al.,Science 376, eabk3112(2022). DOI: 10.1126/science.abk31
- https://www.nobelprize.org/prizes/medicine/2009/illustrated-information/
- https://www.lescienze.it/news/2009/10/05/news/i_nobel_per_la_medicina_o_la_fisiologia-573298/
- Kam, M.L.W., Nguyen, T.T.T. & Ngeow, J.Y.Y. Telomere biology disorders. npj Genom. Med.6, 36 (2021). https://doi.org/10.1038/s41525-021-00198-5
- https://www.hgsc.bcm.edu/other-mammals/y-chromosome-genome-project
- https://www.pacb.com/blog/the-evolution-of-dna-sequencing-tools/

Lascia un commento